特別客員教授
 郷 通子(ごう・みちこ)
郷 通子(ごう・みちこ)
Mitiko Go
専門分野/生命情報学、生物物理学、進化学
職位:特別客員教授、名誉教授
学位:理学博士(名古屋大学)
- 名古屋大学大学院理学研究科博士課程修了
- 名古屋大学理学部生物学科教授、東京大学分子細胞生物学研究所客員教授、長浜バイオ大学教授・学部長、お茶の水女子大学学長を歴任。
研究テーマ
(1)選択的スプライシングによるタンパク質の多様化
(2)RNA編集の実態解析とその意義の重要性
(3)モジュールに基づくタンパク質間相互作用のデザイン、モデリングとイントロンの起源など
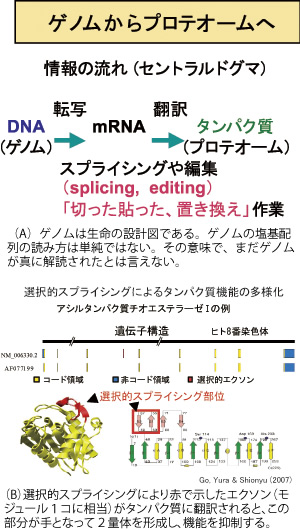
ヒトをはじめとする多様な生物のゲノム情報が急速に集積している。ゲノムの塩基配列に従って、生命現象が展開される。しかし、ゲノムの塩基配列からタンパク質への「翻訳」は、単純ではなく、ある時は塩基配列を読み飛ばしたり(選択的スプライシング)、ゲノムの塩基配列を別の塩基に読み換えてから、タンパク質に翻訳する(RNA編集)という、驚くほど複雑な仕組みが存在する(図A)。これらの現象の生物的戦略の解明とバイオテクノロジーとしての展開を目指して研究を進めている。
ヒトのcDNAの網羅的な解析が進み、選択的スプライシングの実態が姿を現してきた。選択的スプライシングはひとつの遺伝子から多種類のタンパク質を生む分子機構と考えられ、イントロンの役割が、ここに位置づけられる。真核生物はイントロンをもつが、原核生物は例外を除けば、イントロンをもたない。従って、選択的スプライシングは真核生物の特色である。選択的スプライシングにより、タンパク質機能を調節している例(アシルタンパク質チオエステラーゼI)を図Bに示す。
RNA編集は、ゲノムの塩基配列が、そのままタンパク質に翻訳されない、もう一つの例である。ゲノム塩基配列と、成熟mRNA配列が一致せず、酵素がmRNAを特異的に編集する。植物オルガネラのRUBISCO大サブユニットにおけるRNA 編集部位がRUBISCOの立体構造形成に重要な部位であることを見いだし、生物間共生の戦略に関する仮説を提唱した。
| 研究の応用領域 | 産官学連携で求めるパートナー |
|---|---|
| タンパク質のモジュール置換による機能創出のための独自の計算手法の、タンパク質新機能創出やケミカルバイオロジーなどへの応用。 | 「創薬のためのタンパク質モデリングと相互作用の予測」やコンピュータシミュレーションについて連携。 |
Topics of research
Research Subjects
- Protein diversification by alternative splicing
- Role of RNA editing
- Design and modeling of interaction systems based on protein modules and origin of introns, etc.
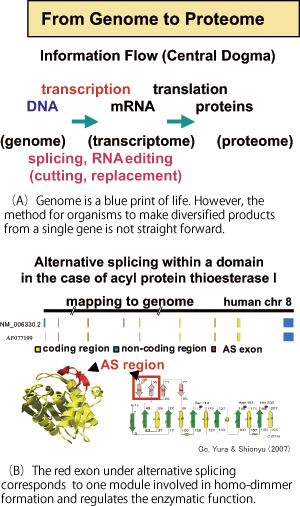
Genomic information of various species is accumulating with high speed. Translation of genomic information to proteins is not straight-forward, and there are unexpectedly complex steps to produce various kinds of proteins from a single gene (alternative splicing ) and translation to a protein after conversion of a single base of mRNA to different kind of base (RNA editing )(Fig.(A)). We analyze these biological strategies to produce diversity and aim to contribute for development of bio-technology.
Extensive analysis of human cDNA reveals the functional role of various alternative splicing (AS). Alternative splicing is a mechanism to produce various proteins from a single gene and this is an important role of introns exiting in eukaryotic genomes. An example of AS regulating the enzymatic function is shown for acyl protein thioesterase I (Fig.(B)).
RNA editing is a mechanism that the base sequence of a genomic DNA is not translated into a protein as it is. Enzymes edit specifically DNA bases to different bases during transcription. RNA editing sites in plant organelles are significantly skewed to residues for protein structural cores. RNA editing seems to be a mechanism to regulate protein function and expression through controlling protein folding.
主な業績論文等
- Mitiko Go, Note: Professor Nobuhiko Saitô’s contribution to statistical mechanics of biopolymers. Biophysics and Physicobiology, Special Issue “Memorial Issue for Prof. Nobuhiko Saitô” 13: 249–250 (2016).
- Shionyu, M., Takahashi, K., Go, M., AS-EAST: a functional annotation tool for putative proteins encoded by alternatively spliced transcripts. Bioinformatics, 28:2076-2077 (2012).
- Hijikata, A., Yura, K., Noguti, T., Go, M., Revisiting gap locations in amino acid swquence alignments and a proposal for a method to improve them by introducing solvent accessibility. Proteins: Structure, Function, and Bioinformatics, 79(6), 1868-1877 (2011).
- Shionyu, M., Yamaguchi, A., Shinoda, K., Takahashi, K., Go, M., AS-ALPS: a database for analyzing the effects of alternative splicing on protein structure, interaction and network in human and mouse. Nucleic Acids Res, 37(Database issue), D305-9 (2009).
- Yura, K., Go, M., Correlation between amino acid residues converted by RNA editing and functional residues in protein three-dimensional structures in plant organelles, BMC Plant Biol Jul 16; 8:79 (2008).






