フロンティアバイオサイエンス学科
 白井 剛(しらい・つよし)
白井 剛(しらい・つよし)
Tsuyoshi Shirai
専門分野/情報構造生物学研究キーワード/タンパク質工学、ドラッグデザイン
職位:研究科長、教授
学位:博士(理学)(名古屋大学)
- 名古屋大学大学院理学研究科生物学専攻退学
- 名古屋大学大学院工学研究科助手、生物分子工学研究所主任研究員を歴任
研究テーマ
情報生物学( バイオインフォマティクス) と、X線結晶解析実験による構造生物学を手段として、両者を統合したタンパク質・プロテオームの機能・構造の解析手法を研究しています。
1)超分子モデリングパイプラインの開発
Protein Data Bankの構造情報を分類整理した2次データベースはこれまで人力により構築されていましたが、これをパーソナルコンピュータで完全自動で行うタンパク質立体構造2次データベース構築システム(SIRD) を開発しました(http://sird.nagahama-i-bio.ac.jp/sird/)。このシステムを使えば、未知の超分子複合体(分子が集合した生体巨大分子)の構造を予測することができます。現在、共同研究者とともに創薬等支援技術基盤プラットフォーム事業「超分子モデリングパイプラインの構築」によりこのシステムを、分子間相互作用標的ドラッグデザインを支援する、世界初の超分子モデリングパイプラインに成長させる為の研究を行っています。
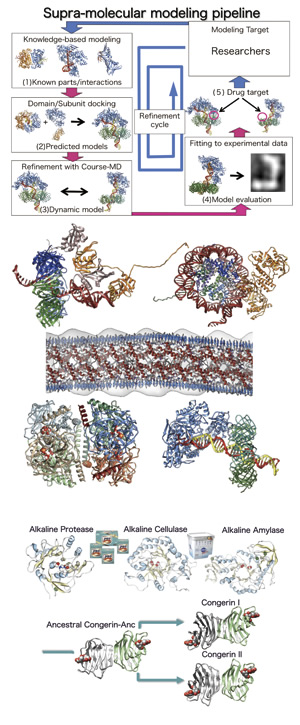
2)超分子構造生物学
SIRDなどの計算機資源を利用して、主として共同研究により、実験的な構造解析の困難なタンパク質複合体や超分子複合体の計算機モデリングを実践しています。狂牛病の原因となるプリオンタンパク質の繊維構造、DNA複製を行う超分子であるレプリソームの構成要素(PCNA-DNA-DNAポリメラーゼ、DNAリガーゼ、ヌクレアーゼなど)の構造、あるいは糖代謝酵素複合体の分子モデリングを行い、機能解析・相互作用解析・実験デザインなどに応用しています。
3)産業用酵素・祖先型タンパク質などの構造解析研究
家庭用洗剤などには、アルカリ性環境などに耐性を持つ酵素が利用されています。プロテアーゼ、セルラーゼ、アミラーゼなど実際に産業利用されている酵素の構造をX線結晶解析により明らかにし、アルカリ耐性の生じるメカニズムを提唱しました。また深海細菌の酵素のX線結晶解析から、高圧力耐性の解明も進めています。さらに、情報生物学はタンパク質の大昔の姿( 祖先タンパク質) の構造を再現する為に使うことができます。魚類の生体防御タンパク質コンジェリンの祖先遺伝子配列を計算し、実際に作成した祖先タンパク質の全体構造を解析することに世界で初めて成功しました。
| 研究の応用領域 | 産官学連携で求めるパートナー |
|---|---|
| タンパク質工学・ドラッグデザイン・データベース・情報生物学解析システム開発 | 酵素・タンパク質関連企業 医薬品関連企業 IT関連企業 |
Topics of research
We are developing the methods in analyzing protein/proteome system by combining the bioinformatics and the experimental, mainly X-ray crystallography, approaches.
1) Development of Supra-molecular Modeling Pipeline
The determined structures of proteins in the Protein Data Bank have been conventionally classif ied manually by expert researchers. In order to cope with the rapidly inf lating database, the fully-automated 2ry PDB processing system (SIRD) has been developed through our research (http://sird.nagahama-i-bio.ac.jp/sird/). This system can be used to predict unknown structures of biological supra-molecules (large functional assemble of bio-molecules in organisms). Under a collaboration with my colleagues, the SIRD system is further developed into supra-molecular modeling pipeline, which will be the f irst integrated computational resource for modeling biological supra-molecules and assistingstructurebased drug designs targeted to macromolecular interactions, with the support from Platform for Drug design, Informatics, and Structural lifescience (PDIS)project.
2) Computer-aided modeling of supramolecular complexes
By using SIRD and other developed methods, practical modeling of protein complexes and supra-molecules has been executed, mainly under collaboration with researchers in this and other institutes. For example, the computational models for prion f iber (responsible factor of BSE), parts of replisome (dynamic supra-molecular complex required for DNA replication), and complexes of carbohydrate metabolizing enzymes have been constructed, and used for predicting functions or interactions, and for designing experiments.
3) Structural analyses of industrial enzymes, and ancestral proteins
The enzymes that are resistant to extreme conditions such as high-alkaline or high-temperature, are industrially used as the builder of detergents. The structures of detergent builder protease, cellulase, and amylase were determined with X-ray crystallography, and the molecular mechanisms for alkaline-adaptation of proteins was proposed. Also the glucosidase from the deepest-sea bacteria was analyzed to understand protein adaptation to a high-pressure.The bioinformatics techniques can be used to calculate ancient structure of genes/proteins. The ancestral sequence of f ish congerin (innate-immune protein) was predicted by using computer. The ancestral congeringene was synthesized, and its three-dimensional structure was determined as the f irst time for a full-length ancestral protein.
主な業績論文等
- Hijikata A, Tsuji T, Shionyu M, Shirai T, Decoding disease-causing mechanisms of missense mutations from supramolecular structures, Sci. Rep. 7, 8541 (2017)
- Shionyu-Mitsuyama C, Hijikata A, Tsuji T, Shirai T, Classification of ligand molecules in PDB with graph match-based structural superposition. J. Struct. Funct. Gen., 17, 135-146 (2016)
- Nakae S, Hijikata A, Tsuji T, Yonezawa K, Kouyama K, Mayanagi K, Ishino S, Ishino Y, Shirai T, Structure of EndoMS-DNA complex as mismatch-restriction endonuclease. Structure, 24, 1960-1971 (2016)
- Tsuji T, Yoda T, Shirai T, Deciphering Supramolecular Structures with Protein-Protein Interaction Network Modeling. Sci. Rep. 5, 16341 (2015)
- Shirai T, Saito M, Kobayashi A, Asano M, Hizume M, Ikeda S, Teruya K, Morita M, Kitamoto T, Evaluating prion models on comprehensive mutation data of mouse PrP, Structure, 22, 560-571 (2014)






