フロンティアバイオサイエンス学科
 大森 義裕(おおもり・よしひろ)
大森 義裕(おおもり・よしひろ)
Yoshihiro Omori
専門分野/ゲノム科学、発生遺伝学、神経科学研究キーワード/進化、遺伝性疾患、ゲノム編集、観賞魚、眼の発生
職位:教授
学位:博士(医学)(東京大学)
- 京都大学大学院工学研究科合成・生物化学専攻修士課程修了
- 東京大学医学研究科医学博士、ハーバード大学医学部(マサチューセッツ眼科耳鼻科病院)博士研究員、財団法人大阪バイオサイエンス研究所副部長、大阪大学蛋白質研究所准教授(理学研究科准教授兼任)を経て本学へ
研究テーマ
近年、次世代DNAシーケンサー技術の発展とゲノム編集技術の発達に伴って、メジャーなモデル生物以外のもつ生物学的な特性を研究することが可能となりつつある。キンギョはフナを原種とする観賞魚であるが、デメキンやランチュウなどの様々な形態や色を持つ品種が存在し、現在、世界で70以上の品種が飼育されている(図Omori & Kon JB 2018より改変)。これらの中には、眼球や骨格の形態など発生生物学的に興味深い表現型を持つものや、網膜変性疾患や骨形成異常などヒト疾患と関連する異常を持つものも多い。私たちは、米国NIHとの国際共同研究により、世界に先駆けてキンギョの全ゲノムを解読した。このことでキンギョの表現型研究の扉が開かれた。私たちの研究室では、以下のような研究をすすめている。

1)様々なキンギョ品種を用いた脊椎動物の形態形成メカニズムとヒト疾患発症機構の理解
キンギョの様々な品種を掛け合わせて雑種交配を行い、これらの子孫のゲノムDNAを次世代DNAシーケンサーを使って解析し、高性能コンピューターにより計算することで、様々なキンギョ品種の特徴を生み出す遺伝子の仕組みを明らかにすることができる。これには、ゲノムワイド関連解析(GWAS)と呼ばれる手法を用いる。遺伝子の同定後は、ゲノム編集が容易な熱帯魚のゼブラフィッシュを使って遺伝子の機能をより詳しく解析する。キンギョは私たちヒトと同じ脊椎動物であり、病気の発症機構は類似していることが多い。これらの研究により、発生生物学的に重要な分子メカニズムの解明や、ヒト疾患の発症機構の解明に繋げることができる。
2)フナの祖先種における全ゲノム重複と遺伝子進化の研究
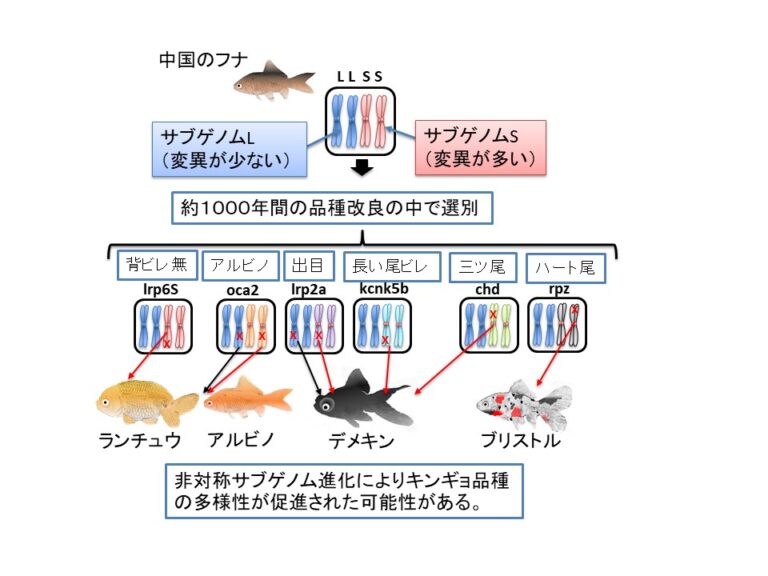
キンギョの原種であるフナの祖先種では1400万年前に全ゲノム重複と呼ばれるすべての遺伝子が倍加する現象が起こったことが、キンギョのゲノム解析により明らかとなった。私たちヒトを含む脊椎動物でも約5億年前に全ゲノム重複を経験したことがわかっており、キンギョの遺伝子を研究することで脊椎動物の進化の中で起こった全ゲノム重複のメカニズムを明らかにすることができる。私たちは、全ゲノム重複後の1400万年の間に非対称サブゲノム進化と呼ばれる特殊な変化がキンギョのゲノムに起こっていることを発見しており、その仕組みについて研究を進めている。また、コイやフナが進化の中で獲得した特性を環境適応進化の観点から研究を行っている。
| 研究の応用領域 | 産官学連携で求めるパートナー |
|---|---|
| 医薬品開発、食品産業、ペット産業、水産育種、環境関連ビジネス等 | 医薬品・食料品関連企業、大学・自治体の研究機関 |
Topics of research
The recent development of next-generation DNA sequencing (NGS) and genome editing technologies has enabled us to study the biological characteristics of non-model organisms. Goldfish (Carassius auratusus) are ornamental teleost fish that were originally domesticated from wild goldfish in China. At least 70 goldfish strains are currently being produced, with phenotypically diverse body shapes, colorations, scales, and fin-, eye-, and hood morphologies that include biologically interesting phenotypes. Certain goldfish strains express phenotypes that are similar to those in some human diseases. Therefore, goldfish serve as suitable model organisms for understanding the molecular basis of vertebrate development and evolution and human diseases. We recently established a high-quality goldfish genome sequence using long-read sequencing technology (Chen & Omori et al., 2019). This investigation provides new tools for the genetic analysis of various goldfish phenotypes. In our laboratory, we focus on the following projects:
1) Understanding the molecular basis of vertebrate development and evolution, as well as the pathogenesis of human diseases, by studying the various phenotypes expressed by goldfish strains.
To elucidate the molecular mechanisms underlying goldfish phenotypes, we crossbreed various goldfish strains and use NGS to analyze the genomic DNA sequences of the offspring. We then use a genome-wide association analysis (GWAS) and functional genome approach that includes bulk/single-cell RNA-seq and ATAC-seq. The gene functions are elucidated using transgenic and genome-edited zebrafish.
2) Whole-genome duplication (WGD) and gene evolution in wild goldfish and crucian carp ancestors.
Genomic analyses of goldfish revealed that a WGD event (duplication of all genes in the genome) occurred approximately 14 million years ago in a goldfish ancestor genome. Two rounds of WGD occurred approximately 500 million years ago in vertebrates, including humans. We aim to clarify the mechanism of vertebrate gene evolution post WGD by analyzing gene evolution in the goldfish genome. Additionally, we also study environmental adaptations in wild goldfish and crucian carp.
主な業績論文等
- Kon T, Omori Y#, Fukuta K, Wada H, Watanabe M, Chen Z, Iwasaki M, Mishina T, Matsuzaki SS, Yoshihara D, Arakawa J, Kawakami K, Toyoda A, Burgess SM, Noguchi H, Furukawa T The genetic basis of morphological diversity in domesticated goldfish. Current Biology 2020;30(12):2211-2418 #corresponding author.
- Chen, Z.*, Omori Y.*, Koren, S., Shirokiya, T., Kuroda, T., Miyamoto, A., Wada, H., Fujiyama, A., Toyoda, A., Zhang, S., Wolfsberg, T.G., Kawakami, K., Phillippy, A.M., Mullikin, J.C., and Burgess, S.M. De Novo assembly of the goldfish (Carassius auratus) genome and the evolution of genes after whole-genome duplication. Science Advances, 2019;5(6) eaav0547. *equally contributed
- Omori Y, Kubo S, Kon T, Furuhashi M, Narita H, Kominami T, Ueno A, Tsutsumi R, Chaya T, Yamamoto H, Suetake I, Ueno S, Koseki H, Nakagawa A, Furukawa T, Samd7 is a cell type-specific PRC1 component essential for establishing retinal rod photoreceptor identity. Proc Natl Acad. Sci. USA 2017 114(39):E8264-E8273
- Omori Y, Chaya T, Katoh K, Kajimura N, Sato S, Muraoka K, Ueno S, Koyasu T, Kondo M, Furukawa T. Negative regulation of ciliary length by ciliary male germ cell-associated kinase (Mak) is required for retinal photoreceptor survival. Proc Natl Acad Sci U S A. 2010, 107(52):22671-22676.
- Sato S, Omori Y, Katoh K, Kondo M, Kanagawa M, Miyata M, Funabiki K, Koyasu T, Kajimura N, Miyoshi T, Sawai H, Kobayashi K, Tani A, Toda T, Usukura J, Tano Y, Fujikado T and Furukawa T, Pikachurin, a dystroglycan ligand, is essential for photoreceptor ribbon synapse formation. Nature Neuroscience 2008, 11(8)923-931
- Omori Y, Zhao C, Saras A, Mukhopadhyay S, Kim W, Furukawa T, Sengupta P, Veraksa A, Malicki J. Elipsa is an early determinant of ciliogenesis that links the IFT particle to membrane-associated small GTPase Rab8. Nature Cell Biology. 2008, 10(4):437-44.






